时光匆匆,暑假也过半了吧?大家都趁着暑假出去游山玩水了,小编看也是跟着在朋友圈云旅游了。假期虽好,不要忘记读文献哦,悄悄进步,开学惊艳实验室的其他小伙伴,嘻嘻~~
《QKI shuttles inteRNAl m7G-modified transcripts into stress granules and modulates mRNA metabolism》2023年7月20发布在Cell 上,新鲜出炉、影响因子64.5,让我们膜拜一下高分文章都是如何搭建文章框架的。内容比较多,满满都是干货,我们闲话少说,开始学习吧~~QKI、m7G、SG文章开头作者就锁定了m7G修饰,使用LC-MS/MS分析了不同类型RNA中m7G的丰度,包括总RNA、小RNA(17-200nt)、rRNA、 mRNA。在HeLa细胞和 HepG2细胞中,m7G/G水平在总RNA中约为0.1%,在小RNA(主要是tRNAs)中约为1%,在RNA和帽缺失的polyA+mRNA中约为0.03%(图1B)。且在两种细胞类型中,细胞质mRNA的内部m7G/G水平都显著高于核mRNA(图1C),这意味着内部m7G修饰可能在细胞质mRNA代谢中发挥作用。METTL1或WDR4缺失显著降低了小RNA和帽缺失polyA+ mRNA的m7G丰度(图1D-1G)。相反,野生型(WT) METTL1过表达显著提高了小RNA和帽缺polyA+ mRNA中的m7G/G水平,过表达WDR4也明显增加了内部m7G修饰(图1H-1J)。
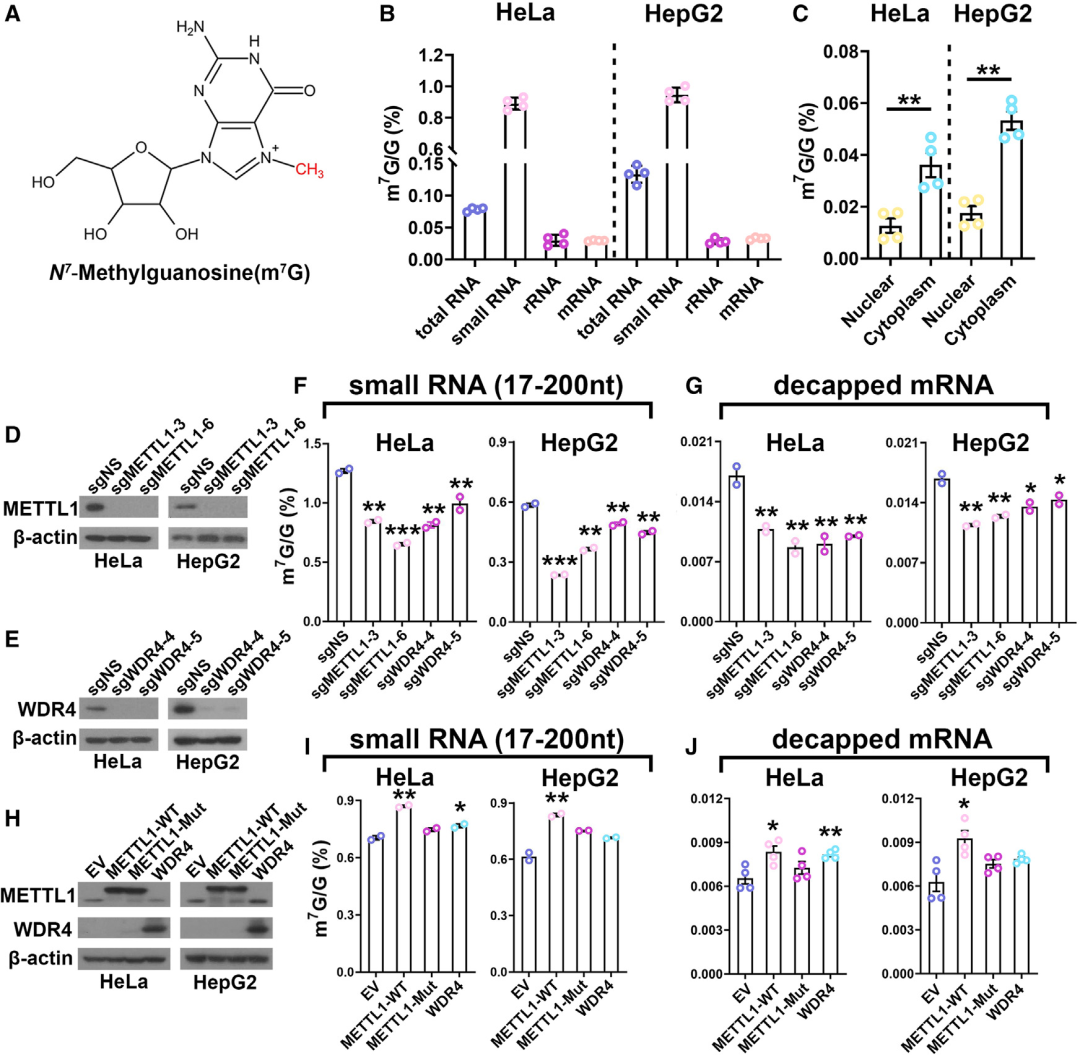
Fig 2 QKIs是内部mRNA m7G的结合蛋白
为了鉴定直接与内部m7G修饰结合的蛋白质,作者采用RNA亲和层析,使用内部m7G修饰的寡聚物诱饵或未修饰的对照结合质谱进行无偏倚筛选。鉴于m7G修饰的RNA寡核苷酸尚未上市,作者合成了两个60-mer的寡核苷酸,其中GTP或m7GTP作为单个G位点的独特来源(图2A和2B)。质谱结果显示,一些蛋白质在含m7G的RNA/蛋白复合物中选择性富集,而不是含G的复合物(图2C)。进一步的RNA下拉实验证实,m7G诱饵富集的蛋白质,如LRPPRC、IGF2BP1和QKI,确实对内部m7G修饰的线性RNA具有更高的结合能力(图2D-2E)。据报道内部m7G的mRNA在富含GA的环境中富集,作者推测其甲基转移酶METTL1和真正的结合蛋白应该优先结合富含GA的基序。正如预期的那样,METTL1的首要基序是富含GA的基序“UGAAGA”(图2F)。有趣的是,QKI还识别出了一个富含GA的基序“GAUGAU”(图2F)。无细胞RNA下拉实验显示,所有三种重组QKI蛋白,QKI5、QKI6和QKI7,比未修饰的蛋白更优先地与m7G修饰的寡核苷酸结合(图2G)。QKI蛋白在体外也特异性识别m7G修饰的寡核苷酸(图2H)。与此一致的是,无细胞RNA免疫沉淀(RIP)和体外RIP检测均显示QKI5 /6/7结合mRNA中内部m7G显著富集(图2I和2J)。总的来说,这些结果表明QKI选择性地识别内部m7G修饰的RNA。接下来,作者用m7G MeRIP-seq分析了mRNA内部m7G甲基化组,并在HepG2细胞中确定了QKIs与RIP-seq的转录组范围内的结合位点。帽缺失mRNA的m7G MeRIP-seq显示,m7G峰在起始密码子区域富集(图2K), m7G峰显著富集“GANGAN (N = a /C/U/ G)”基序(图2L)。当将QKI RIP-seq数据与m7G MeRIP-seq数据整合时,大约30%的内部m7G修饰转录物与QKI5、QKI6和QKI7的假定靶点重叠(图2M)。QKIs的经典RNA识别元件(RREs)为“ACUAAY”或“YUAAY”(Y = C/U)。RIP-seq中,QKI5、QKI6和QKI7的首要基序一致为UUAAUA (QKI5)或ACUAAC (QKI6和QKI7)(图2N)。第二丰富的QKI基序“GANGA (N = A/C/U/G)”与内部m7G基序“GANGAN”高度相似(图2N)。特别是QKI7 “GANGAN”基序与内部m7G基序相同(图2L-2N)。更重要的是,在整个转录组水平上,m7G峰的分布与QKI结合峰有很好的重叠(图2O)。这些数据表明,除了保守的经典RREs外,QKI还识别mRNA中的内部m7G基序。METTL1缺失整体抑制了QKI7结合峰,其低结合峰(4723)是超结合峰(2167个)的两倍(图2P和2Q)。综上所述,我们的研究结果表明,METTL1负责mRNA内部m7G甲基化,QKIs在体外和体外专门识别mRNA 内部m7G修饰。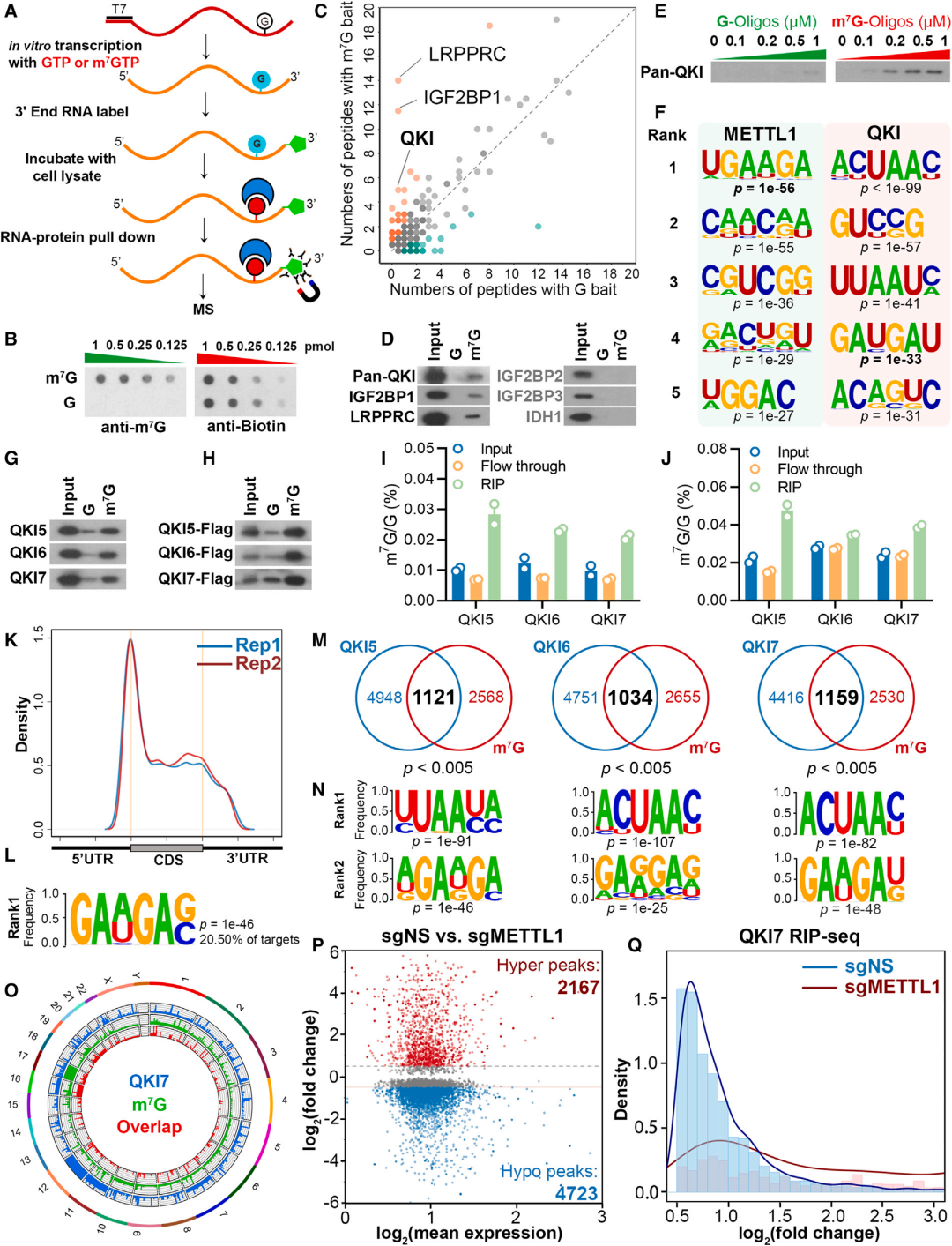
Figure 3 QKI6和QKI7在应激条件下与G3BP1结合
QKI5、QKI6和QKI7的细胞定位有很大差异。QKI5主要位于细胞核中,而QKI6和QKI7同时位于细胞核中,特别是在细胞核中(图3A-3C)。最近的研究表明,mRNA内部的m7G是动态调控的,在应激条件下可能是mRNA翻译的关键调控因子,但其潜在机制尚不清楚。应激适应对细胞的存活至关重要。许多应激条件抑制了mRNA的整体翻译并诱导SG的形成。为了研究QKI是否与核心SG蛋白相互作用并调控内部m7G修饰mRNA,对蛋白-蛋白相互作用数据库GeneMANIA进行分析,发现QKI与多个核心SG蛋白密切相互作用,包括G3BP1、PTBP1、HNRNPK等(图3D)。COIP实验显示,QKI6在正常和应激条件下与G3BP1结合,而QKI7仅在应激条件下与G3BP1相互作用(图3E和3F)。此外,原位邻位连接分析(PLA)证实SGs中QKI6/QKI7和G3BP1之间的物理相互作用(图3G和3H)。免疫荧光(IF)染色再次证实,QKI6/7主要与G3BP1共定位于胞质SGs,而QKI5仅定位于细胞核(图3I-3K)。QKI6-RFP和QKI7-RFP融合蛋白在SGs中也明显与G3BP1- GFP共定位(图3L和S3E-S3H)。进一步构建6个截断的QKI蛋白,发现C端,特别是D2片段(氨基酸268-312)对于与G3BP1的相互作用至关重要(图3M)。综上所述,在应激条件下,QKI6和QKI7直接与SG核心蛋白G3BP1相互作用,并在SGs内共定位,这意味着QKI可能在应激条件下参与mRNA代谢。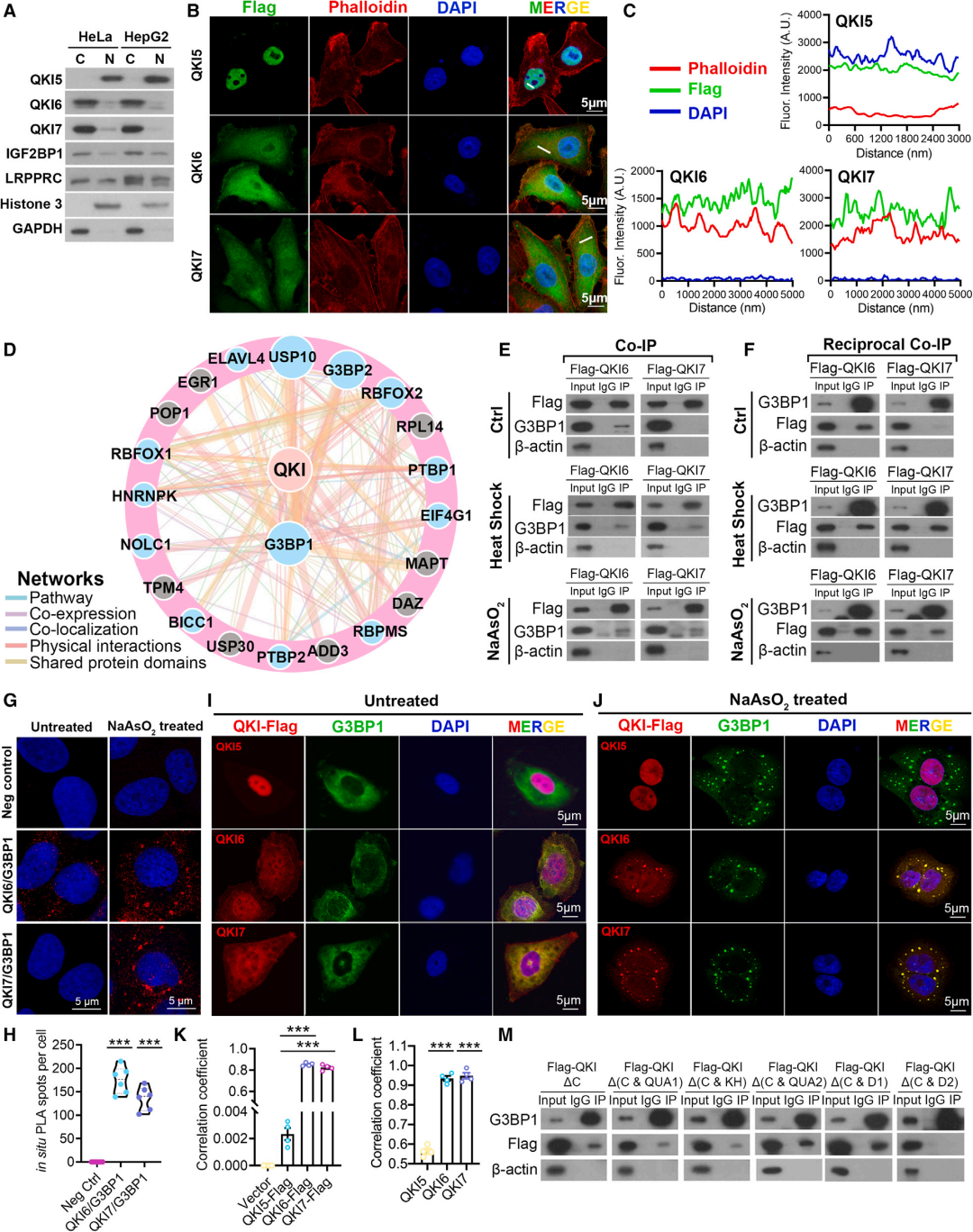
Figure 4 QKI7在应激条件下,将内部m7G修饰的mRNA穿梭到SGs中
对应激条件下的HeLa和U2OS细胞进行polyA信号和内部m7G信号成像,探索QKI是否将内部m7G修饰的mRNA转运到SG细胞中(图4A)。虽然在正常情况下,这两种信号在细胞质中都是弥漫性的,但在应激条件下它们在SGs中的积累(图4B)。定量分析表明,SGs在应激条件下内部m7G比polyA信号更丰富(图4C),表明内部m7G修饰的mRNA优先被招募到SGs中。通过评估不溶性RG、细胞质和细胞核中帽缺失的mRNA的内部m7G水平进一步验证了这一点,RG中mRNA具有更高的内部m7G水平,特别是在应激下(图4D)。METTL1 KO显著减轻了SGs内部的m7G信号,而WT METTL1的过表达恢复了m7G信号(图4E-4G),表明METTL1甲基转移酶活性是SGs内部m7G修饰mRNA富集的必要条件。此外,QKIs的缺失或过表达并没有显著改变帽缺失mRNA的内部m7G水平或METTL1和G3BP1的表达水平(图4H-4J)。亚砷酸钠处理后SGs的内部m7G水平因为QKI缺失显著降低(图4K)。此外,QKI KO介导的SGs内部m7G的减少通过WT QKI7的加入部分恢复,但没有恢复不能与内部m7G结合的截断QKI(图4L-4N)。QKI KO显著降低了富含SG的polyA+mRNA的整体内部m7G丰度,而QKI7过表达时则相反(图4O)。总之,研究结果表明,QKI7可以识别胞质内m7G修饰的mRNA,并在应激条件下将它们运送到SGs中。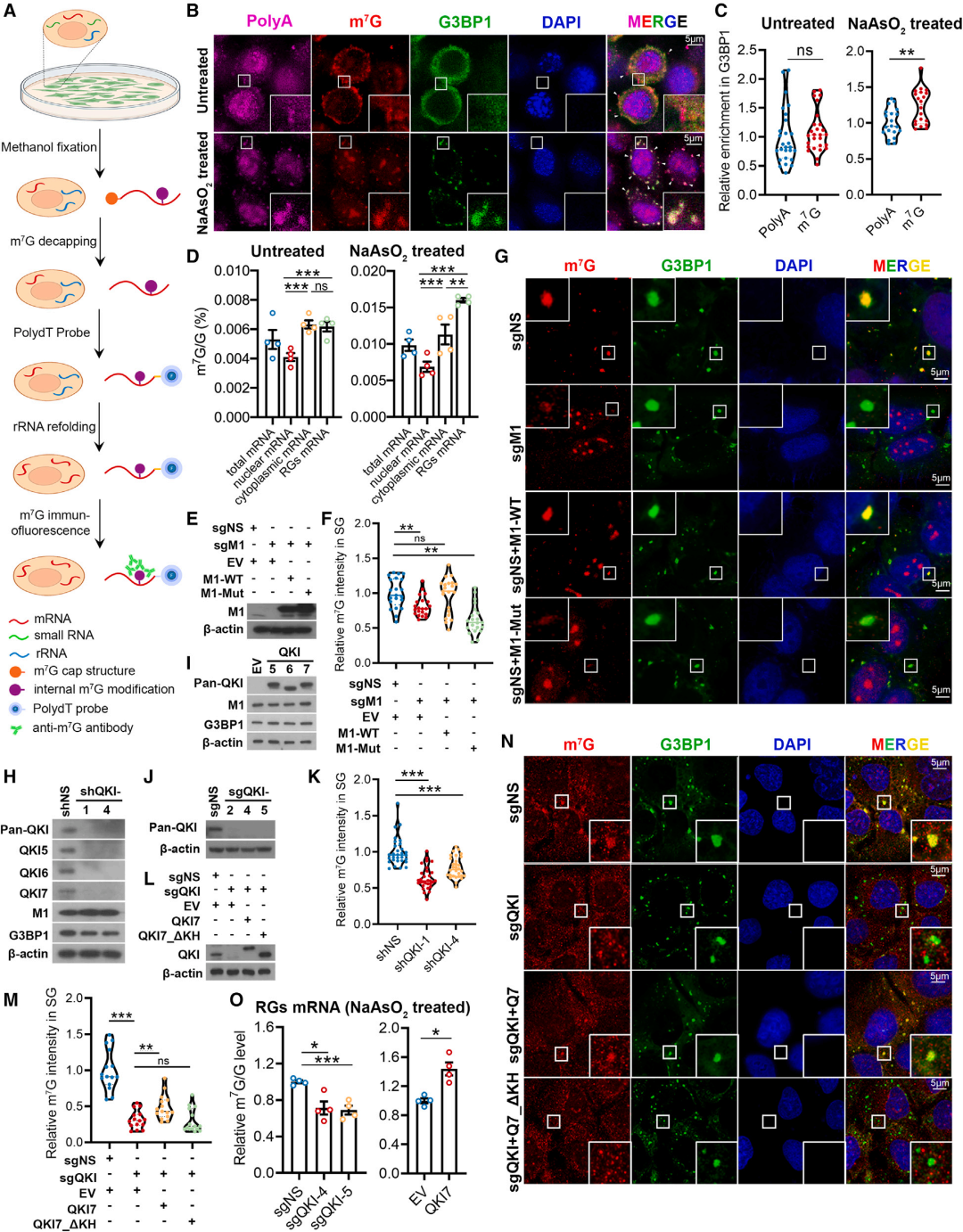
Fig 5 SGs中QKI7结合以及内部m7G修饰mRNA的全转录组分析
为了全面研究QKI7对富含SG的mRNA代谢的影响,作者在U2OS细胞中使用了QKI7 RIP-seq和m7G MeRIP-seq发现,在U2OS细胞中,QKI7也同时具有神经节内部m7G基序和“ACUAAC”典型RRE基序,并且内部m7G修饰在神经节环境中显著富集(图5A)。进一步将测序数据与来自U2OS细胞的SG RNA-seq数据集进行了整合,发现RIP-seq检测到63.4%的SG富集基因是QKI7结合的转录本,而只有13.8%的SG缺失基因属于QKI7结合的mRNA(图5C和5D),与之前研究一致,即QKI7结合的转录本在SGs中高度富集。QKI7结合和内部m7G修饰转录本中富集SG的基因的百分比远远高于SG缺失的基因(图5E)。进一步的通路富集分析显示,在U2OS细胞中,Hippo信号通路和癌症通路中富集了“m7G修饰和QKI7结合基因”和“m7G修饰和QKI7结合和SG富集基因”(图5F)“m7G修饰、QKI7结合和SG富集的转录本”中有11个和17个分别属于Hippo信号通路和癌症通路的核心富集基因(图5G)。然后,作者评估了QKI7过表达或KO后U2OS细胞中不溶性SG富集组分中这些核心富集基因的丰度(图5H和5I)。正如预期的那样,在28个核心富集基因中,当QKI7过表达时,23个基因在SGs中显著富集;而当QKI7缺失时,在SGs中有16个基因缺失(图5J)。因此,这16个基因被认为是“SG反应基因”。QKI7可能作为一个驱动因素,协调m7G修饰的转录本被招募到SGs中,从而调节应激条件下的细胞活性。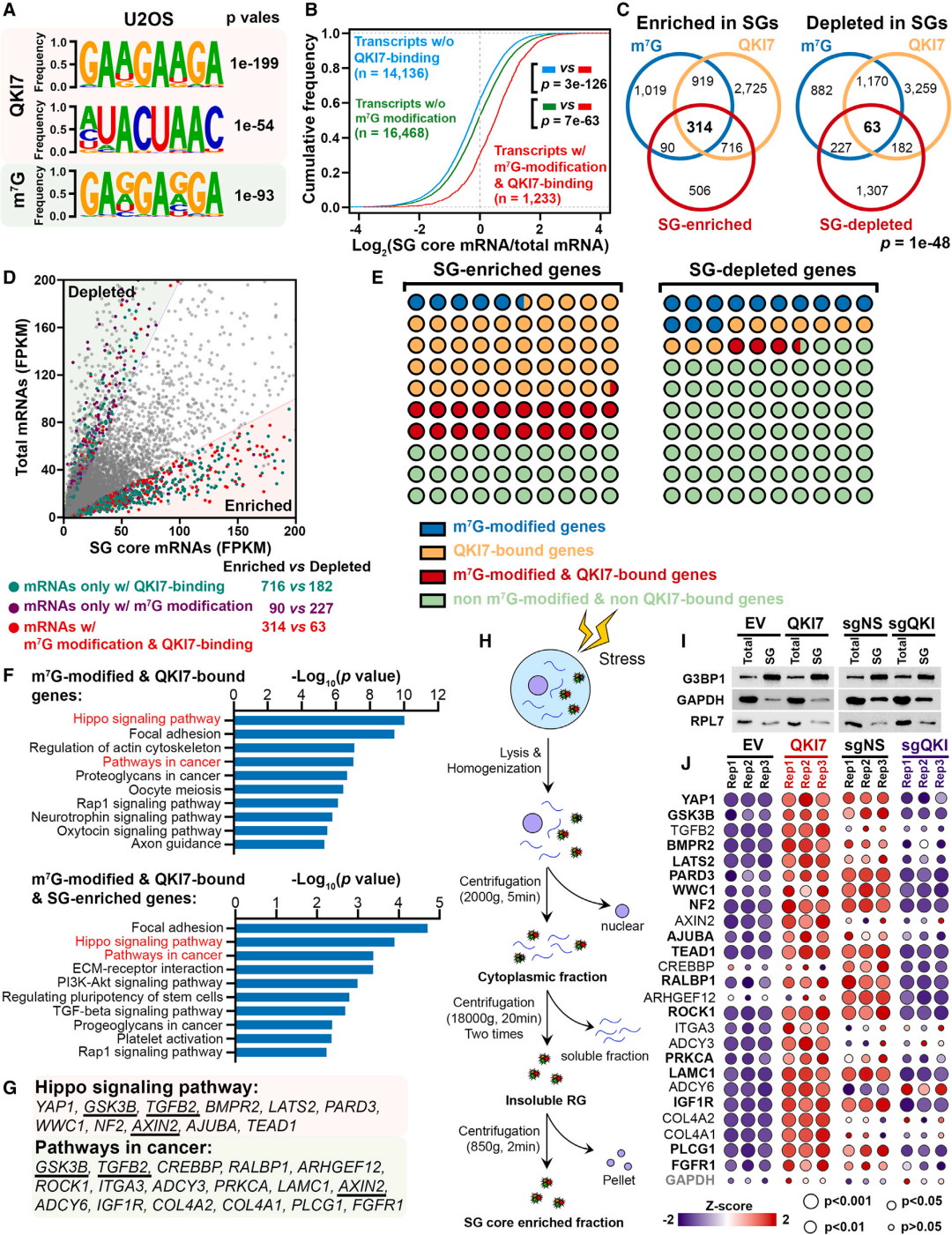
Fig 6 在应激条件下QKI7调节一系列内部m7G修饰mRNA的稳定性和翻译效率
大量研究表明,暴露于应激环境后,RBP可以调节多种细胞质过程,如RNA稳定性、RNA存储、翻译效率和定位于细胞质颗粒。上述研究已经证明了内部m7G修饰的mRNA在SGs中高度积累,接下来评估QKI是否可以调节这些m7G修饰的mRNA在SGs中的命运。mRNA稳定性分析发现QKI KO显著降低了mRNA的整体半衰期,而过表达WT QKI7,而不是截断的QKI7,可以在很大程度上挽救这些表型(图6A)。这些潜在的靶基因与QKI7结合的转录本(图6B)和富含SG的转录本(图6C)重叠良好。QKI可以调节这些代表性的SG应答基因的半衰期(图6D),这意味着QKI7过表达可以将这些基因穿梭储存到SGs中,提高它们在逆境条件下的稳定性。然而,当分析QKI在不同多聚体中的分布时,在正常情况下,QKI7保留在游离核糖核蛋白中;而在应激条件下,QKI7转移到40S和60S核糖体亚基部分(图6E),在那里形成凝聚翻译起始复合物。这些数据表明,尽管QKI7对全局蛋白合成的影响轻微,但它可能潜在地调节一系列靶mRNA的翻译效率,特别是在胁迫条件下。RNA-seq和核糖核酸-seq评估应激条件下QKI7对翻译效率的影响,发现过表达QKI7显著降低了“QKI7结合转录本”和“SG富集转录本”的翻译效率(图6F和6G)。为了确定QKI-m7G-SG轴的翻译依赖的直接mRNA靶点,作者主要关注Hippo信号通路和/或癌症通路中的候选靶点,因为这两条通路是富含SG的m7G修饰和QKI7结合基因的最高富集通路(图5F和5G)。在16个已验证的SG应答基因中确定了5个候选靶基因,包括GSK3B、TEAD1、ROCK1、IGF1R和BMPR2,这些基因在QKI7过表达时可能会降低翻译效率(图6H-6J)。在应激条件下,QKI7过表达显著降低了除BMPR2外这些候选靶mRNA在多聚体组分中的富集(图6H-6I),支持QKI7隔离其mRNA靶点并在压力下抑制其翻译的观点。此外,METTL1缺失显著抑制了QKI7与这些转录本的结合(图6K)。进一步研究发现,在亚砷酸钠处理后,QKI7过表达显著降低了GSK3B、TEAD1、ROCK1和IGF1R的蛋白水平和翻译效率,但没有降低它们的mRNA水平(图6L)。研究结果表明,内部m7G结合蛋白QKI7通过将一系列m7G修饰的转录本隔离在SGs中来微调它们在胁迫下的稳定性和/或翻译效率(图6M)。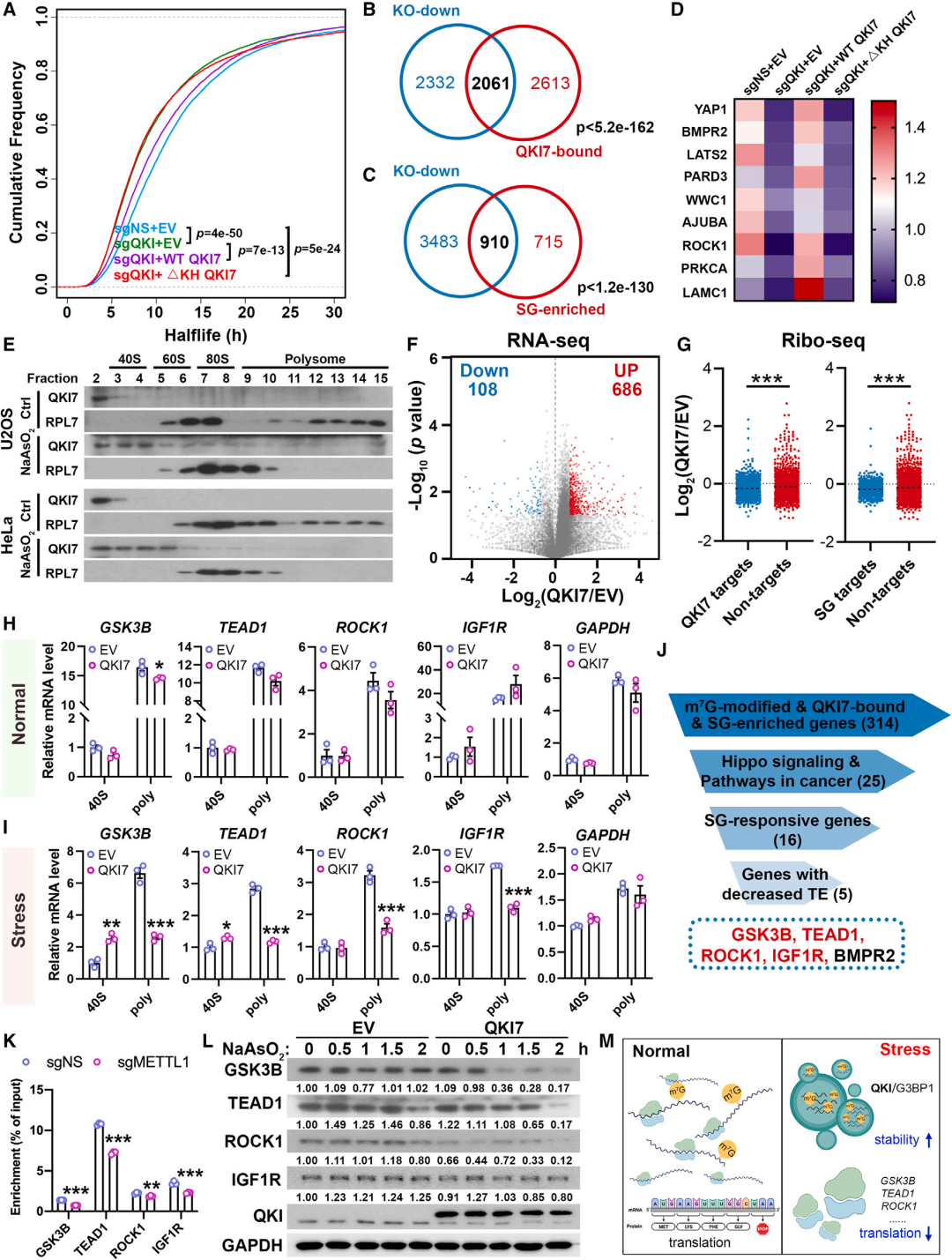
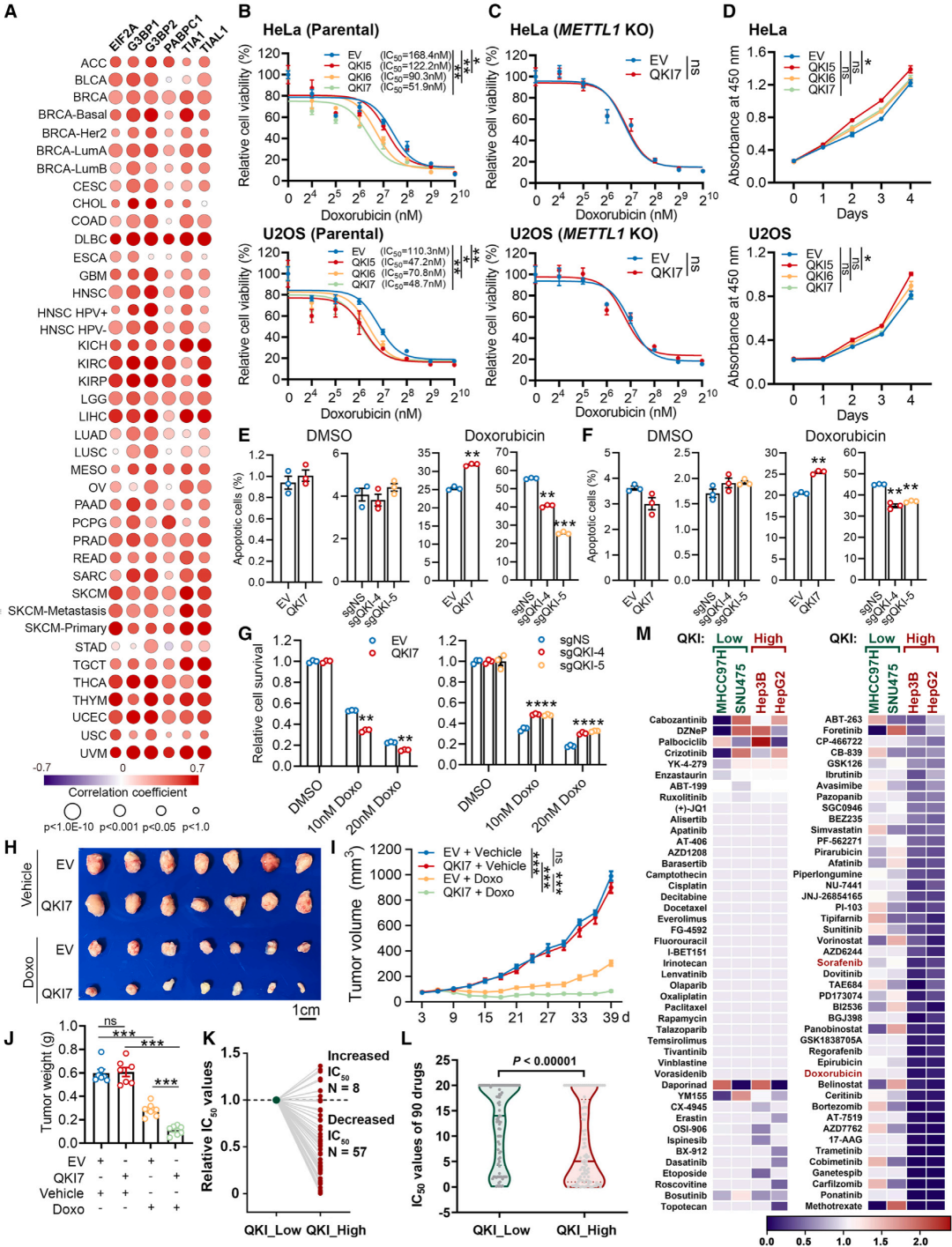
癌症基因组图谱(TCGA)数据集显示,QKI和SG核心成分在不同癌症类型中的表达呈显著正相关(图7A),这意味着QKI可能在癌症中与SG相关的生物过程中发挥广泛作用。过表达QKIs显著降低了亲代癌细胞中阿霉素的IC50值,但在METTL1 KO细胞中没有(图7B和7C),这表明METTL1是QKI诱导的癌细胞对阿霉素致敏所必需的。过表达QKIs并不能显著影响癌细胞的增殖(图7D),提示QKIs介导的化疗应答与细胞增殖的改变无关。相反,QKI7的过表达和缺失分别显著促进和抑制了阿霉素诱导的癌细胞凋亡(图7E和7F)。克隆生存试验也显示,QKI7过表达和KO分别增强和抑制癌细胞对阿霉素的敏感性(图7G)。体内异种移植模型证实,QKI7过表达不影响肿瘤增殖,但显著增强肿瘤对阿霉素治疗的敏感性(图7H-7J)。Hippo信号通路已成为多种肿瘤耐药的关键,而GSK3B是化疗耐药的关键介质。在阿霉素诱导的应激条件下,QKI7过表达显著降低了Hippo信号通路中两个核心富集基因GSK3B和TEAD1的蛋白水平(图6J)。表明QKI7 - m7G轴介导的Hippo信号通路的沉默可能有助于提高癌细胞对化疗的敏感性。对肝癌药物基因组景观研究数据集分析还显示,与QKI7低表达的肝癌细胞系(MHCC97H和SNU475)相比,肝癌HKI7高表达(HepG2和Hep3B)对各种化疗或靶向治疗药物更为敏感(图7K-7M)。总的来说,mRNA内部m7G修饰结合蛋白QKI7可使各种癌细胞对化疗药物(如阿霉素和索拉非尼)敏感。
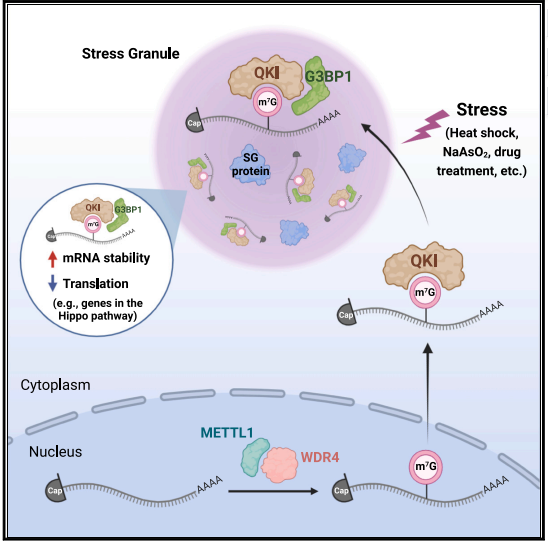
 结合技术路线图来看,作者报道了内部m7G修饰的mRNA被Quaking protein (QKIs)选择性识别。通过转录组分析/绘制内部m7G甲基组和QKI结合位点,确定了1000多个具有保守的“GANGAN (N = a /C/U/G)”基序以及高置信度m7G修饰的和QKI结合的mRNA靶标。QKI7(C端)与应激颗粒(SG)核心蛋白G3BP1相互作用,并将内部m7G修饰的转录物转运到SGs中,以调节mRNA在应激条件下的稳定性和翻译。具体来说,QKI7降低了Hippo信号通路中必需基因的翻译效率,调节靶mRNA代谢和细胞耐药性。
结合技术路线图来看,作者报道了内部m7G修饰的mRNA被Quaking protein (QKIs)选择性识别。通过转录组分析/绘制内部m7G甲基组和QKI结合位点,确定了1000多个具有保守的“GANGAN (N = a /C/U/G)”基序以及高置信度m7G修饰的和QKI结合的mRNA靶标。QKI7(C端)与应激颗粒(SG)核心蛋白G3BP1相互作用,并将内部m7G修饰的转录物转运到SGs中,以调节mRNA在应激条件下的稳定性和翻译。具体来说,QKI7降低了Hippo信号通路中必需基因的翻译效率,调节靶mRNA代谢和细胞耐药性。
综上,本月“神刊文献解读”就到此为止啦。我们下个月再见~~


 2023-08-01 16:00:47
2023-08-01 16:00:47






 结合技术路线图来看,作者报道了内部m7G修饰的mRNA被Quaking protein (QKIs)选择性识别。通过转录组分析/绘制内部m7G甲基组和QKI结合位点,确定了1000多个具有保守的“GANGAN (N = a /C/U/G)”基序以及高置信度m7G修饰的和QKI结合的mRNA靶标。QKI7(C端)与应激颗粒(SG)核心蛋白G3BP1相互作用,并将内部m7G修饰的转录物转运到SGs中,以调节mRNA在应激条件下的稳定性和翻译。具体来说,QKI7降低了Hippo信号通路中必需基因的翻译效率,调节靶mRNA代谢和细胞耐药性。
结合技术路线图来看,作者报道了内部m7G修饰的mRNA被Quaking protein (QKIs)选择性识别。通过转录组分析/绘制内部m7G甲基组和QKI结合位点,确定了1000多个具有保守的“GANGAN (N = a /C/U/G)”基序以及高置信度m7G修饰的和QKI结合的mRNA靶标。QKI7(C端)与应激颗粒(SG)核心蛋白G3BP1相互作用,并将内部m7G修饰的转录物转运到SGs中,以调节mRNA在应激条件下的稳定性和翻译。具体来说,QKI7降低了Hippo信号通路中必需基因的翻译效率,调节靶mRNA代谢和细胞耐药性。
