

ChIP实验中引物序列的选择策略
 2024-11-14 11:28:12
2024-11-14 11:28:12
来源/作者:普拉特泽-生物医学整体课题外包平台
在Chromatin Immunoprecipitation(ChIP)实验中,引物序列的选择是实验成功的关键步骤之一。ChIP技术是一种研究蛋白质与DNA相互作用的重要方法,它通过特定的抗体将结合在DNA上的蛋白质“拉下来”,随后利用PCR和测序等技术鉴定这些被绑定的DNA区域。
选择合适的引物序列不仅能提高实验的灵敏度和特异性,还能确保结果的准确性和可靠性。分子检测平台为广大科研实验人员提供ChIP实验实验服务,先一起来学习学习ChIP实验中选择合适引物序列的详细策略。

一、基于目标基因启动子区域的设计
ChIP实验主要检测转录因子与基因启动子区的结合情况,因此引物设计应优先选择目的基因的启动子区域。启动子区域通常位于转录起始位点上游1000bp至2000bp的DNA片段内,因为这是转录因子结合的主要区域。不过,也有文献报道转录因子可能结合在更上游或下游的位置,但为了确保实验的普遍性和可靠性
选择转录起始位点上游2000bp的片段作为引物设计区域是一个较为稳妥的选择。
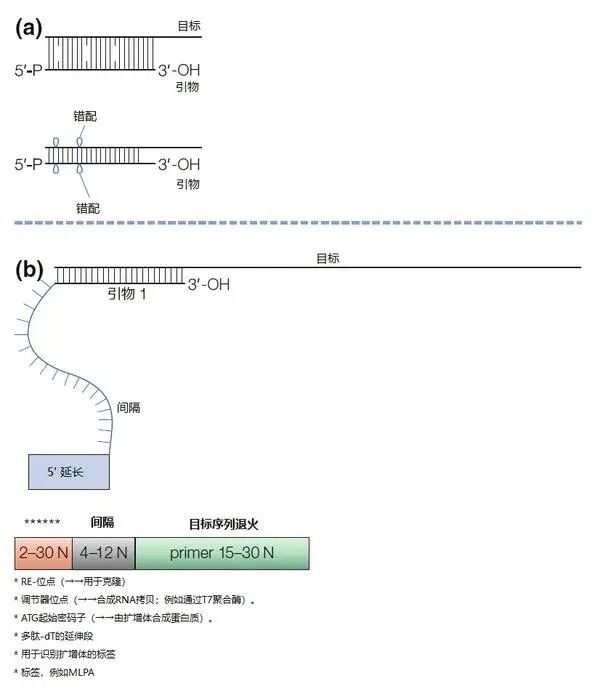
二、引物设计的基本原则
▲引物长度:引物长度一般为15-30bp,常用的是18-27bp,最长不应超过38bp。引物过短可能导致非特异性扩增,而过长则可能在引物内部形成二级结构,如发夹环,影响PCR扩增效率。
▲GC含量:引物的GC含量一般在40%-60%之间,过高或过低都不利于PCR扩增。GC含量过低可能导致扩增效果不佳,而过高则易出现非特异性条带。
▲熔解温度(Tm值):
引物的熔解温度是指DNA双链解链成为单链的中点温度,对于PCR反应的退火步骤至关重要。引物的Tm值应在55-75℃之间,上下游引物的Tm值差异应不超过2-3℃。通常,可以通过公式Tm=4(G+C)+2(A+T)来计算引物的Tm值。
▲引物序列:引物序列在模板内应当没有相似性较高的序列,尤其是3'端相似性较高的序列,否则容易导致错配。引物3'端出现3个以上的连续碱基(如GGG或CCC)也会增加错误引发的机率。此外,应避免在引物的3'端使用碱基A,因为A的错配效率明显高于其他三个碱基。
▲二级结构:引物设计中应避免出现发夹结构或引物二聚体,这些结构可能导致PCR反应失败。因此,在引物设计时,应确保引物序列的二级结构能值不超过4.5kcal/mol。
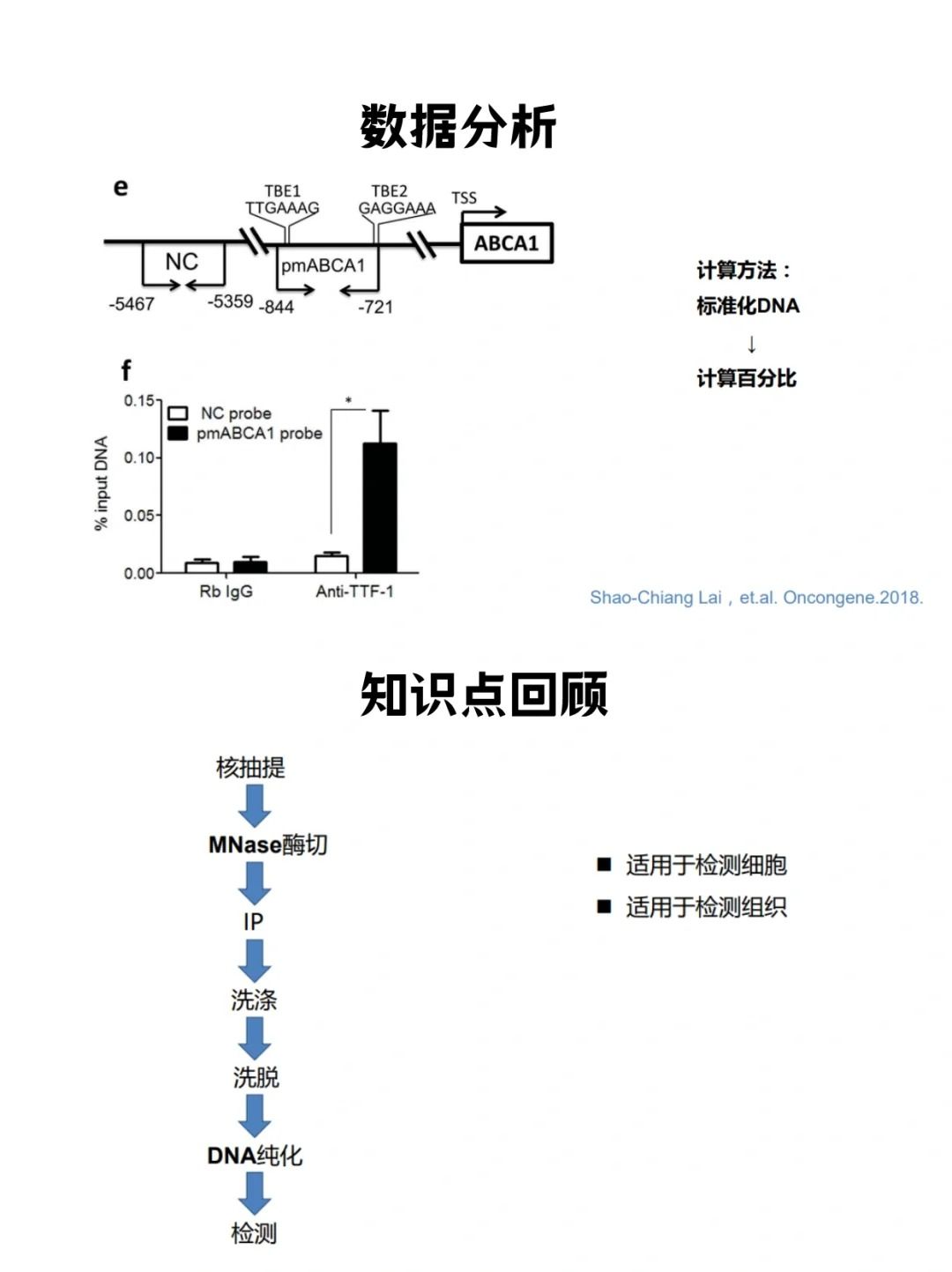
三、结合生物信息学预测优化引物设计
在进行ChIP实验之前,可以利用生物信息学工具如JASPAR、ENCODE等来预测蛋白质可能结合的DNA序列。这些工具基于大量的实验数据和机器学习算法,能够较为准确地预测转录因子的结合位点。
根据预测的结合位点来设计引物,可以确保引物只扩增目标DNA区域,从而提高实验的特异性和灵敏度。
四、实验验证与调整
设计好的引物需要通过实验进行验证。可以在含有目标DNA的模板和不含目标DNA的负对照样本上进行PCR扩增,确保引物的特异性和扩增效率。
如果实验结果不理想,可以根据PCR产物的电泳图谱和测序结果对引物进行微调,如调整引物长度、GC含量或Tm值等,以获得最佳的扩增效果。
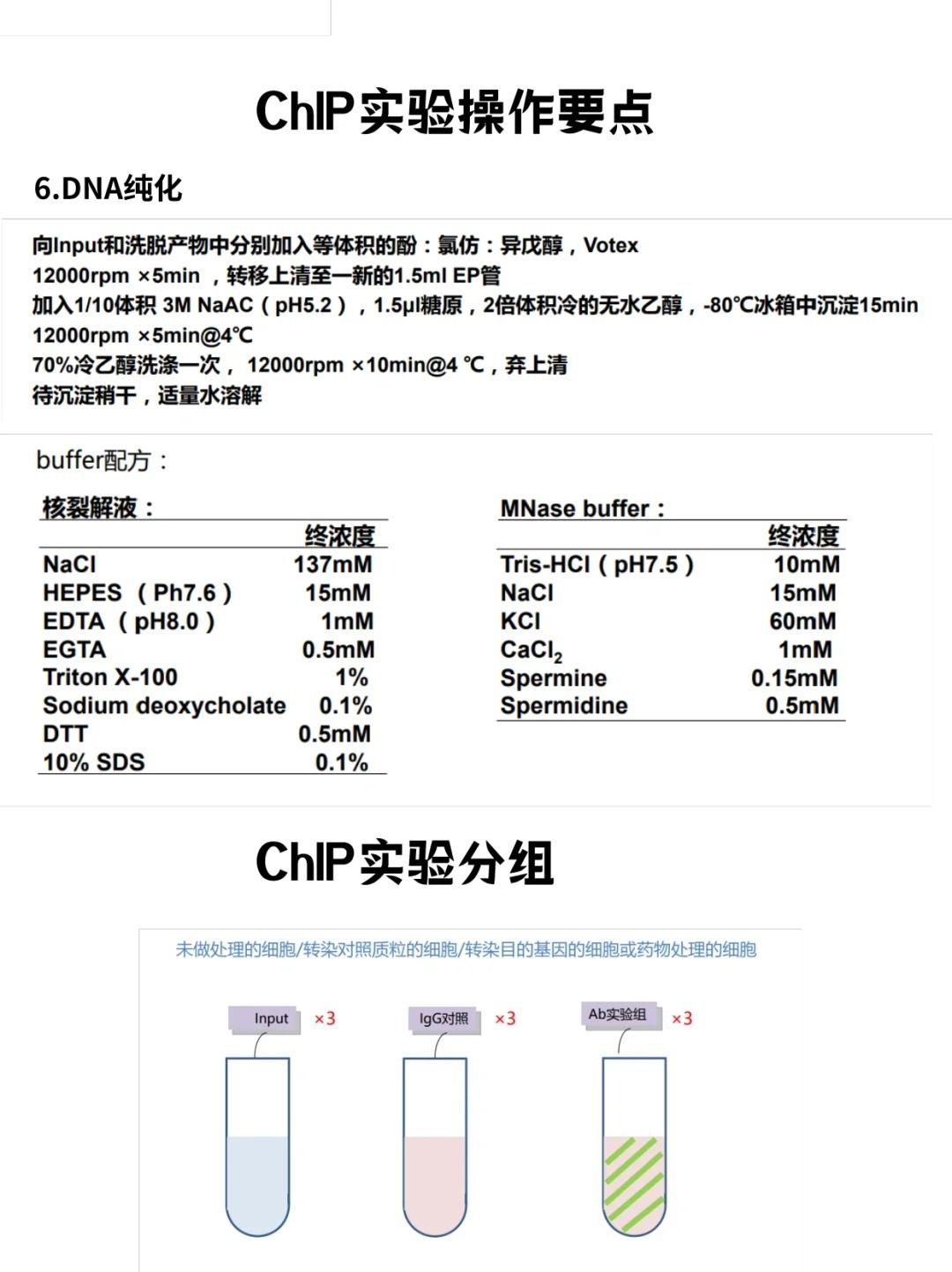
五、注意事项
在设计引物时,应尽可能避免引物自身或引物之间形成连续4个碱基以上的互补序列,以防止它们形成稳定的二聚体。
引物的5'端序列对PCR影响不太大,因此常用来引进修饰位点或标记物,如荧光标记或其他标签,便于扩增子的检测和分析。

在实际设计过程中,往往很难同时满足所有条件,因此只需要在设计引物时尽可能满足主要条件即可。同时,需要注意各种模板的引物设计难度不一,具体情况需要具体看待。
综上所述,在ChIP实验中选择合适的引物序列是一个综合考量的过程,包括目标蛋白质的信息、生物信息学预测、引物设计原则以及实验验证等多个方面。正确的引物设计不仅能够提高实验的灵敏度和特异性,还能确保结果的准确性和可靠性
为后续的转录因子功能研究和表观遗传学研究提供有力的支持。
如果你总是在做实验时总会遇上这样那样的问题需要帮助时,请记得在ChIP实验中遇到技术问题可添加技术微信:18570028002
我司还提供ChIP实验外包服务或实验培训!同时还可以加入我们实验技术交流群哦!

